空间转录组|数据读入,标准数据形式外,还有哪些"天残地缺"可以读取
空间转录组测序可以同时获得细胞的空间位置信息和基因表达数据,虽然囿于当前单个spot的精度问题,但是在组织细胞功能,肿瘤生物学、发育过程等需要空间位置的研究领域仍然可以提供很多非常有价值的东西。
本节会在spaceranger处理fastq,Seurat处理标准数据(h5 spatial文件夹)外,额外提供以下几种情况如何处理
(1)raw/filtered_feature_bc_matrix 的三个文件 spatial 文件夹
(2)矩阵文件 spatial 文件夹
(3)spatial文件夹如果只提供了tissue_hires_image.png
(4)未提供spatial文件夹,只提供了位置信息
数据来源于2022年CELL 文章 A human fetal lung cell atlas uncovers proximal-distal gradients of differentiation and key regulators of epithelial fates 中的空转Fastq数据(E-MTAB-11265)。
一 fastq数据,spaceranger分析
如若初始是fastq数据,首先在10X官网(/cn/support/software/space-ranger/downloads)下载spaceranger软件然后安装,仍然是使用count参数处理fastq 数据即可。
/path/spaceranger count --id=6332STDY10289523 \--transcriptome=/path/refdata-gex-GRCh38-2020-A/ \--fastqs=/path/ST_data/ \--sample=6332STDY10289523 \--image=/path/V10S24-031_D1.jpg \--slide=V10S24-031 \--area=D1
注意slide 和 area的参数 ,可能来源于图片的命名(本示例中的V10S24-031_D1.jpg) ,可能来自于method 或者code的链接中。
如果没有slide信息的话,可以设置为--unknown-slide
二 Seurat 数据读入
上面spaceranger count后会得到outs文件夹,里面有网页版的质控报告,loupe文件,spatial文件夹 和 h5文件等,用于后续分析。
1, Load10X_Spatial 最优输入
outs文件夹中的 filtered_feature_bc_matrix.h5 spatial文件夹 ,注意这两部分结果需要在同一级。
library(Seurat)library(hdf5r)spe = Load10X_Spatial(data.dir = "./outs/",
filename = "filtered_feature_bc_matrix.h5",
assay = "Spatial",
slice = "Lung")head(spe@meta.data,2)#
orig.ident nCount_Spatial nFeature_Spatial#AAACAAGTATCTCCCA-1 SeuratProject
7232
2774#AAACACCAATAACTGC-1 SeuratProject
20915
6318
#空转的话,就是SpatialFeaturePlot ,相较于单细胞转录组的FeaturePlotSpatialFeaturePlot(spe, features = "nFeature_Spatial")
额,报错了,如下:
Error in FUN(left, right) : non-numeric argument to binary operator
推荐google搜索,解决方法如下(将spe@images$Lung@coordinates的信息改为):
for (i in colnames((spe@images$Lung@coordinates))) { spe@images$Lung@coordinates[[i]] <- as.integer(spe@images$Lung@coordinates[[i]])}
再次尝试,问题解决,注意如果你的数据没有该报错可以不用as.integer 这部分!!!
SpatialFeaturePlot(spe, features = "nFeature_Spatial")
p0 <- SpatialDimPlot(spe,alpha = 0)p1 <- SpatialDimPlot(spe,alpha = 1)p0 p1
非常建议通过 str(spe)查看一下空转的数据结构 ,有助于后续更好的分析!
2,10X的三个文件 spatial文件夹
默认的Load10X_Spatial函数不能像单细胞转录组一样直接读取3个文件,通过View(Load10X_Spatial) 发现该函数写的比较死。其实我们只需要提取前面读取单细胞数据部分,使用Read10X即可
spe2 = Read10X("./outs/filtered_feature_bc_matrix/")image2 <- Read10X_Image(image.dir = file.path("./outs/",
"spatial"), filter.matrix = TRUE)spe2 <- CreateSeuratObject(counts = spe2, assay = "Spatial")
image2 <- image2[Cells(x = spe2)]DefaultAssay(spe2 = image2) <- "Spatial"spe2[["slice1"]] <- image2#没有报错,无需转化for (i in colnames((spe2@images$slice1@coordinates))) { spe2@images$slice1@coordinates[[i]] <- as.integer(spe2@images$slice1@coordinates[[i]])}
SpatialFeaturePlot(spe2, features = "nFeature_Spatial")
而Load10X_Spatial函数中的spe2[["slice1"]] 部分,也就是为什么在默认情况下image的slide 名字是slice1 ,所以如果多样本分析的时候记得改名字。
3,读取rds文件
部分文献会提供rds文件,但是因为版本不一致常会遇到问题
spe3 <- readRDS("./STData/P1N_Spatial.rds")
SpatialFeaturePlot(spe3 , features = "MS4A1")
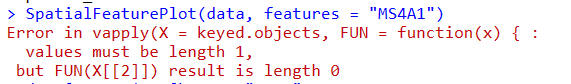
google检索解决方案:https://github.com/satijalab/seurat/issues/6312 ,可以UpdateSeuratObject 一下
spe3<- UpdateSeuratObject(spe3)SpatialFeaturePlot(spe3, features = "MS4A1")
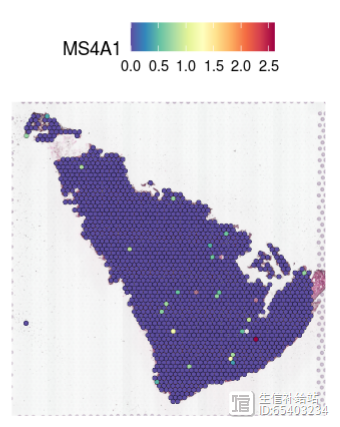
4,只提供 tissue_hires_image.png
spatial文件夹中会有tissue_hires_image.png和 tissue_lowres_image.png 两种tiff图,而Read10X_Image 函数中默认读取 tissue_lowres_image.png图,
因此如果(1)文献只提供了tissue_hires_image.png 或者(2)想绘制高清一些的图,可以使用如下的方法
4.1 尚无Seurat object
文献只提供hires图时候直接使用Load10X_Spatial会报错,可以先指定使用tissue_hires_image图片 ,如下
img = Read10X_Image("./outs/spatial/",
image.name = "tissue_hires_image.png")spe4 = Load10X_Spatial("./outs/", image = img)spe4@images$slice1@scale.factors$lowres = spe4@images$slice1@scale.factors$hires#没有报错,无需转化for (i in colnames((spe4@images$slice1@coordinates))) { spe4@images$slice1@coordinates[[i]] <- as.integer(spe4@images$slice1@coordinates[[i]])}#对比一下两张图的差异p1 <- SpatialFeaturePlot(spe2, features = "nFeature_Spatial")p2 <- SpatialFeaturePlot(spe4, features = "nFeature_Spatial")p1 p2

其他的图可能会差异更明显。
4.2 已有Seurat object
如果前面已经创建了seurat object ,不想从头分析的话 也可以直接替换的,如下先重新定义一个spe5
spe5 <- spe2
lowres = spe5@images$slice1hires = Read10X_Image("./outs/spatial/",
image.name = "tissue_hires_image.png")
hires@scale.factors$lowres = hires@scale.factors$hires#Set the assay and key to the same as the lowres (I'm not sure if this is necessary or not).hires@assay = lowres@assayhires@key = lowres@key#Finally, add the image into the object and everything should be good!spe5@images$slice1 = hires#没有报错,无需转化for (i in colnames((spe5@images$slice1@coordinates))) { spe5@images$slice1@coordinates[[i]] <- as.integer(spe5@images$slice1@coordinates[[i]])}
p1 <- SpatialFeaturePlot(spe2, features = "nFeature_Spatial")p2 <- SpatialFeaturePlot(spe5, features = "nFeature_Spatial")p1 p2
该解答来源于https://github.com/satijalab/seurat/discussions/4833
5,只提供位置信息
没有提供spatial文件夹,只提供了空转spot的位置信息,可以按照单细胞的方式读取,但是无法绘制如上的空转切片为背景的图。
以上是总结的一些空转数据的读入方式,请查收!
◆ ◆ ◆ ◆ ◆
精心整理(含图PLUS版)|R语言生信分析,可视化(R统计,ggplot2绘图,生信图形可视化汇总)
RNAseq纯生信挖掘思路分享?不,主要是送你代码!(建议收藏)
scRNA分析 | 定制 美化FeaturePlot 图,你需要的都在这
单细胞常见的可视化方式有DimPlot,FeaturePlot,DotPlot,VlnPlot和DoHeatmap几种,Seurat中均可以很简单的实现,但是文献中的图大多会精美很多。之前跟SCI学umap图|ggplot2绘制umap图,坐标位置,颜色,大小还不是你说了算介绍过DimPlot的一些调整方法。本文介绍FeaturePlot的美化方式,包含以下几个方面:站长网2023-07-29 16:35:5600050SAP ERP系统SD模块常用增强之二:创建和修改交货单的检查校验
在SAP/ERP项目的实施中销售管理模块(SD)的创建和修改发货单(DN)经常会遇到检查校验的需求,来防止业务人员创建错误的DN,SAP系统这方面的配置功能也非常强大,通常情况下不需要写开发代码,通过配置可以实现大部分需求,但是在实际项目中还是会遇到一些特殊的需求,不能通过配置实现,需要进行增强开发。SAP系统这方面主要增强点有如下两点:增强点1:程序:MV50AFZ1子例程(FORM):站长网2023-07-27 09:37:180002教你如何打开微信隐藏的拍照功能?打开后照片清晰10倍,简单实用
朋友们大家好,我是小俊,今天小俊给大家分享一下微信中隐藏的一个实用功能,好多朋友还不知道,那就是如何使用微信拍出高清美观又好看的照片,既不需要关闭微信,返回到手机界面打开相机,也可以使用美颜滤镜效果,平时我们在使用微信给好友发送照片的时候啊,都是直接打开微信聊天界面,接着点击拍摄按钮,这样呢就可以将照片发送给好友了,但是好友打开之后啊,会发现照片非常模糊,很不清晰!站长网2023-07-28 13:51:590000【美图美文 -茕茕孑立,形影相吊】
【美图美文-茕茕孑立,形影相吊】图文:网络编辑:小丝制作日期:2023.06.04.*****欢迎光临感谢欣赏,小丝祝您欣赏愉快,天天快乐******站长网2023-07-29 09:30:300000顶级小软件!来3个!
hello大家好,这里是日常爆肝更新的老Y工作室。最新发现了几个不错的小软件,抽空在摸鱼的时候测试了下推荐给大家,觉得有用记得点赞支持下呀01、CAD看图一款小巧免费的CAD快速看图工具,可以进行CAD的一些基础操作,比如面积和长度测量、底图灰显、图层功能、数据导出、图数分离、版本转换、提取文字、打散分解等,内置测量报表可以导出excel。下载地址:蓝奏:/idY4X0w8mm6b站长网2023-07-28 08:42:000003